提速1200倍! MIT開發新一代藥物研發AI,吊打舊模型
眾所周知,整個宇宙充滿著無數分子。
這些分子中又有多少具有潛在的類似藥物的特性,可用於開發挽救生命的藥物呢?是百萬級?還是十億級?又或是萬億級?
答案是:10的60次方。
如此龐大的數字,大大延緩了新藥的研發進度,像新冠等快速傳播的疾病,目前遲遲未有特效藥,也是因為分子種類和數量太大,遠遠超出了現有藥物設計模型可以計算的範圍。
MIT的一個研究團隊偏不信這個邪。不算是吧,那把之前的模型加加速總可以吧?
這一加速,就是1200倍。
他們研究了一款名為「EquiBind」的幾何深度學習模型,該模型比之前最快的計算分子對接模型「QuickVina2-W」快了1200倍, 成功地將類藥物分子與蛋白質結合,減少了藥物試驗失敗的機會和成本。
研究論文即將發表在ICML 2022。
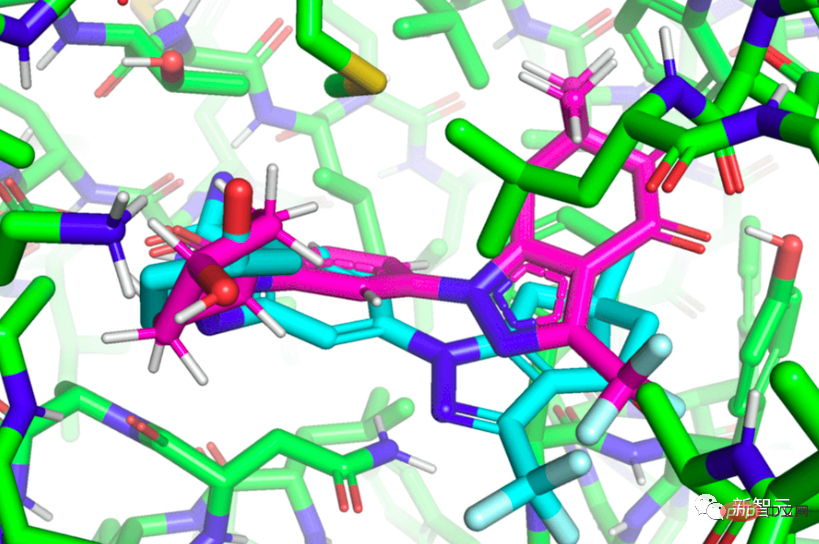
初識「EquiBind」
「EquiBind」是基於前身「EquiDock」研發的,「EquiDock」使用了已故的麻省理工學院AI研究員Octavian-Eugen Ganea開發的技術,去結合兩種蛋白質。 Ganea也是「EquiBind」論文的合著者。
在藥物開發開始之前,研究人員必須找到有前途的藥物樣分子,這些分子可以在藥物發現的過程中正確地結合或「停靠」在特定的蛋白質標靶上。
在與蛋白質成功對接後,結合藥物(配體),可以阻止蛋白質發揮作用。如果這種情況發生在細菌的一種基本蛋白質上,它可以殺死細菌,從而保護人體。
然而,不論是從經濟角度分析,還是從計算角度分析,藥物發現的過程都可能是昂貴的,研發過程動輒耗資數十億美元,並且在FDA最終批准之前,會進行超過十年的開發和測試。
更重要的是,90%的藥物在人體試驗後因為沒有效果或副作用過多而失敗。
所以製藥公司收回這些成本的方法之一就是,提高最終成功研發藥物的價格。
走進「EquiBind」
目前,尋找有希望的候選藥物分子的計算過程是這樣的:大多數最先進的計算模型依賴大量的候選樣本,再加上評分、排名和微調等方法,以獲得配體和蛋白質之間的最佳「匹配」。
Hannes Stärk是MIT電子工程與電腦科學系的一年級研究生,也是這篇論文的主要作者,他把典型的「配體-蛋白質」結合方法比喻為「試圖把鑰匙放進有很多鑰匙孔的鎖中」。

典型的模型在選擇最佳模型之前會花費時間對每個「適合度」進行評分。相較之下,「EquiBind」無需事先了解蛋白質的目標口袋,只需一步就能直接預測出精確的關鍵位置,稱為「盲對接」。
與大多數需要多次嘗試才能找到配體在蛋白質中的有利位置的模型不同,「EquiBind」已經具有內建的幾何推理功能,可幫助模型學習分子的潛在物理特性,並成功地進行歸納。以便在遇到新的或無法識別的資料時成功泛化,以做出更好的預測。
這些發現的發布迅速引起了業內專業人士的注意,其中包括Relay Therapeutics首席數據官Pat Walters。
Walters建議,研究小組可以在一種現有的用於肺癌、白血病和胃腸道腫瘤的藥物和蛋白質上去嘗試他們的模型。儘管大多數傳統的對接方法都未能成功地結合這些蛋白質上的配體,但「EquiBind」成功了。
Walters說:「EquiBind為對接問題提供了一種獨特的解決方案,它結合了姿態預測和結合位點識別。」
「並且這種方法利用了來自數千個公開的晶體結構的訊息,有可能會以新的方式影響該領域。」
Stärk表示:「當其他所有方法都完全錯誤或僅有一個正確時,我們很驚訝,因為EquiBind能夠把它放進正確的口袋,看到這個結果,我們是十分激動的!」
助力「EquiBind」
儘管「EquiBind」已經收到了大量的來自業內專業人士的回饋,而這些回饋幫助了團隊考慮計算模型的實際用途,但Stärk仍希望在7月即將舉行的ICML上找到不同的觀點。
Stärk表示:「我最期待的回饋是關於如何進一步改進該模型的建議。」
「我想和這些研究人員討論,告訴他們我認為下一步可以怎麼做,並鼓勵他們繼續前進,將模型用於他們自己的論文和方法中。目前已經有很多研究人員聯繫了我們,詢問我們這個模型是否會對他們的問題有用。」
此外,這篇文章同樣也是為了紀念Octavian-Eugen Gane,他為幾何機器學習研究做出了至關重要的貢獻,並慷慨地指導了許多學生,他是個有著謙遜靈魂的傑出學者。
今年上半年,他在一次健行中永遠離開了我們。
以上是提速1200倍! MIT開發新一代藥物研發AI,吊打舊模型的詳細內容。更多資訊請關注PHP中文網其他相關文章!

熱AI工具

Undresser.AI Undress
人工智慧驅動的應用程序,用於創建逼真的裸體照片

AI Clothes Remover
用於從照片中去除衣服的線上人工智慧工具。

Undress AI Tool
免費脫衣圖片

Clothoff.io
AI脫衣器

Video Face Swap
使用我們完全免費的人工智慧換臉工具,輕鬆在任何影片中換臉!

熱門文章

熱工具

記事本++7.3.1
好用且免費的程式碼編輯器

SublimeText3漢化版
中文版,非常好用

禪工作室 13.0.1
強大的PHP整合開發環境

Dreamweaver CS6
視覺化網頁開發工具

SublimeText3 Mac版
神級程式碼編輯軟體(SublimeText3)
 全球最強開源 MoE 模型來了,中文能力比肩 GPT-4,價格僅 GPT-4-Turbo 的近百分之一
May 07, 2024 pm 04:13 PM
全球最強開源 MoE 模型來了,中文能力比肩 GPT-4,價格僅 GPT-4-Turbo 的近百分之一
May 07, 2024 pm 04:13 PM
想像一下,一個人工智慧模型,不僅擁有超越傳統運算的能力,還能以更低的成本實現更有效率的效能。這不是科幻,DeepSeek-V2[1],全球最強開源MoE模型來了。 DeepSeek-V2是一個強大的專家混合(MoE)語言模型,具有訓練經濟、推理高效的特點。它由236B個參數組成,其中21B個參數用於啟動每個標記。與DeepSeek67B相比,DeepSeek-V2效能更強,同時節省了42.5%的訓練成本,減少了93.3%的KV緩存,最大生成吞吐量提高到5.76倍。 DeepSeek是一家探索通用人工智
 Google狂喜:JAX性能超越Pytorch、TensorFlow!或成GPU推理訓練最快選擇
Apr 01, 2024 pm 07:46 PM
Google狂喜:JAX性能超越Pytorch、TensorFlow!或成GPU推理訓練最快選擇
Apr 01, 2024 pm 07:46 PM
谷歌力推的JAX在最近的基準測試中表現已經超過Pytorch和TensorFlow,7項指標排名第一。而且測試並不是JAX性能表現最好的TPU上完成的。雖然現在在開發者中,Pytorch依然比Tensorflow更受歡迎。但未來,也許有更多的大型模型會基於JAX平台進行訓練和運行。模型最近,Keras團隊為三個後端(TensorFlow、JAX、PyTorch)與原生PyTorch實作以及搭配TensorFlow的Keras2進行了基準測試。首先,他們為生成式和非生成式人工智慧任務選擇了一組主流
 AI顛覆數學研究!菲爾茲獎得主、華裔數學家領銜11篇頂刊論文|陶哲軒轉贊
Apr 09, 2024 am 11:52 AM
AI顛覆數學研究!菲爾茲獎得主、華裔數學家領銜11篇頂刊論文|陶哲軒轉贊
Apr 09, 2024 am 11:52 AM
AI,的確正在改變數學。最近,一直十分關注這個議題的陶哲軒,轉發了最近一期的《美國數學學會通報》(BulletinoftheAmericanMathematicalSociety)。圍繞著「機器會改變數學嗎?」這個話題,許多數學家發表了自己的觀點,全程火花四射,內容硬核,精彩紛呈。作者陣容強大,包括菲爾茲獎得主AkshayVenkatesh、華裔數學家鄭樂雋、紐大電腦科學家ErnestDavis等多位業界知名學者。 AI的世界已經發生了天翻地覆的變化,要知道,其中許多文章是在一年前提交的,而在這一
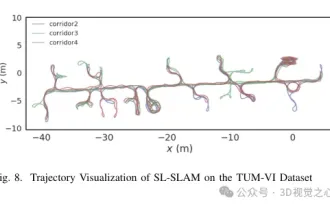 超越ORB-SLAM3! SL-SLAM:低光、嚴重抖動和弱紋理場景全搞定
May 30, 2024 am 09:35 AM
超越ORB-SLAM3! SL-SLAM:低光、嚴重抖動和弱紋理場景全搞定
May 30, 2024 am 09:35 AM
寫在前面今天我們探討下深度學習技術如何改善在複雜環境中基於視覺的SLAM(同時定位與地圖建構)表現。透過將深度特徵提取和深度匹配方法相結合,這裡介紹了一種多功能的混合視覺SLAM系統,旨在提高在諸如低光條件、動態光照、弱紋理區域和嚴重抖動等挑戰性場景中的適應性。我們的系統支援多種模式,包括拓展單目、立體、單目-慣性以及立體-慣性配置。除此之外,也分析如何將視覺SLAM與深度學習方法結合,以啟發其他研究。透過在公共資料集和自採樣資料上的廣泛實驗,展示了SL-SLAM在定位精度和追蹤魯棒性方面優
 你好,電動Atlas!波士頓動力機器人復活,180度詭異動作嚇到馬斯克
Apr 18, 2024 pm 07:58 PM
你好,電動Atlas!波士頓動力機器人復活,180度詭異動作嚇到馬斯克
Apr 18, 2024 pm 07:58 PM
波士頓動力Atlas,正式進入電動機器人時代!昨天,液壓Atlas剛「含淚」退出歷史舞台,今天波士頓動力就宣布:電動Atlas上崗。看來,在商用人形機器人領域,波士頓動力是下定決心要跟特斯拉硬剛一把了。新影片放出後,短短十幾小時內,就已經有一百多萬觀看。舊人離去,新角色登場,這是歷史的必然。毫無疑問,今年是人形機器人的爆發年。網友銳評:機器人的進步,讓今年看起來像人類的開幕式動作、自由度遠超人類,但這真不是恐怖片?影片一開始,Atlas平靜地躺在地上,看起來應該是仰面朝天。接下來,讓人驚掉下巴
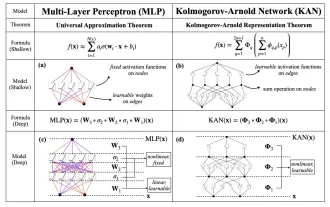 替代MLP的KAN,被開源專案擴展到卷積了
Jun 01, 2024 pm 10:03 PM
替代MLP的KAN,被開源專案擴展到卷積了
Jun 01, 2024 pm 10:03 PM
本月初,來自MIT等機構的研究者提出了一種非常有潛力的MLP替代方法—KAN。 KAN在準確性和可解釋性方面表現優於MLP。而且它能以非常少的參數量勝過以更大參數量運行的MLP。例如,作者表示,他們用KAN以更小的網路和更高的自動化程度重現了DeepMind的結果。具體來說,DeepMind的MLP有大約300,000個參數,而KAN只有約200個參數。 KAN與MLP一樣具有強大的數學基礎,MLP基於通用逼近定理,而KAN基於Kolmogorov-Arnold表示定理。如下圖所示,KAN在邊上具
 特斯拉機器人進廠打工,馬斯克:手的自由度今年將達到22個!
May 06, 2024 pm 04:13 PM
特斯拉機器人進廠打工,馬斯克:手的自由度今年將達到22個!
May 06, 2024 pm 04:13 PM
特斯拉機器人Optimus最新影片出爐,已經可以在工廠裡打工了。正常速度下,它分揀電池(特斯拉的4680電池)是這樣的:官方還放出了20倍速下的樣子——在小小的「工位」上,揀啊揀啊揀:這次放出的影片亮點之一在於Optimus在廠子裡完成這項工作,是完全自主的,全程沒有人為的干預。而且在Optimus的視角之下,它還可以把放歪了的電池重新撿起來放置,主打一個自動糾錯:對於Optimus的手,英偉達科學家JimFan給出了高度的評價:Optimus的手是全球五指機器人裡最靈巧的之一。它的手不僅有觸覺
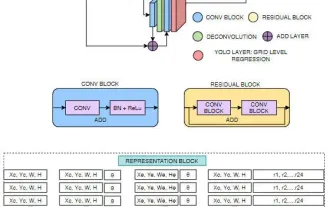 FisheyeDetNet:首個以魚眼相機為基礎的目標偵測演算法
Apr 26, 2024 am 11:37 AM
FisheyeDetNet:首個以魚眼相機為基礎的目標偵測演算法
Apr 26, 2024 am 11:37 AM
目標偵測在自動駕駛系統當中是一個比較成熟的問題,其中行人偵測是最早得以部署演算法之一。在多數論文當中已經進行了非常全面的研究。然而,利用魚眼相機進行環視的距離感知相對來說研究較少。由於徑向畸變大,標準的邊界框表示在魚眼相機當中很難實施。為了緩解上述描述,我們探索了擴展邊界框、橢圓、通用多邊形設計為極座標/角度表示,並定義一個實例分割mIOU度量來分析這些表示。所提出的具有多邊形形狀的模型fisheyeDetNet優於其他模型,並同時在用於自動駕駛的Valeo魚眼相機資料集上實現了49.5%的mAP






