
圖0
#不同種類細胞中基因組構象的差異決定了基因表現的特異性,進而決定不同細胞類型的功能差異。長久以來,從原位雜交到高通量檢測如 Hi-C、micro-C 技術,基因組構象檢測的實驗方法通常耗時耗力、成本高且有很強的技術限制。這些方法極大地限制了這些實驗技術在基因組構象研究領域的廣泛應用,尤其是研究稀有細胞類型以及需要大規模驗證基因組構象調控的因果關係等方面。這些方法的限制也長期限制三維基因組構象調控領域的新發現。

圖1
#2023 年1 月9 日,紐約大學醫學院(NYU Grossman School of Medicine)Aristotelis Tsirigos 實驗室和博德研究所(Broad Institute of MIT and Harvard)夏波實驗室合作在Nature Biotechnology 上發表文章《Cell type-specific prediction of 3D chromatin organization enables high-throughput in silico genetic screening》。

#論文網址:https://www.nature.com/articles/s41587-022-01612-8
這項研究中,第一作者紐約大學醫學院博士生譚濟民與夏波博士首先提出了新型多模態機器學習模式C.Origami 來預測特定細胞類型的染色質構象,並基於遺傳篩選的原理提出了全新的高通量計算遺傳篩選(in silico genetic screening, ISGS) 方法,用於鑑定細胞類型特異性的功能基因組元件,協助發現新的染色質構象調控機制。
#圖2
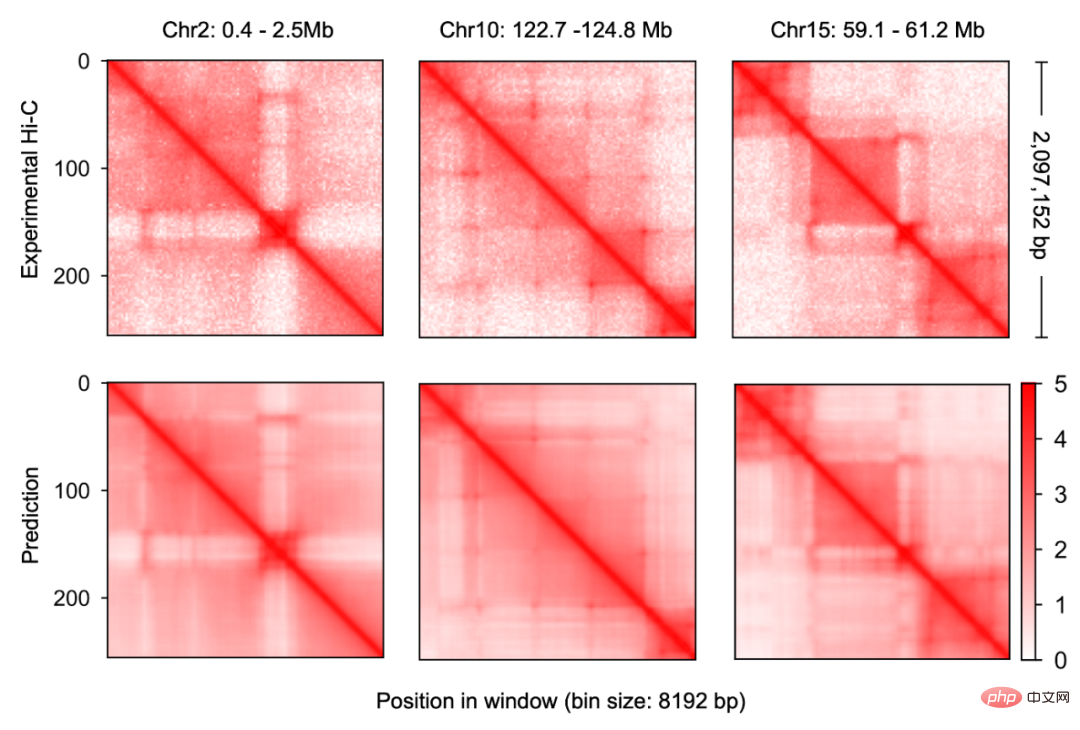
#研究者#首先建構了應用於基因組數據的新型多模態深度學習框架,Origami,使其能有效整合DNA 序列資訊以及細胞特異性的功能基因組信息,進而預測新的基因組資訊。透過反覆調試及模型訓練,研究者發現整合DNA 序列、CTCF 結合狀態(CTCF ChIP-seq)、及ATAC-seq 訊號作為輸入資訊可以準確預測染色質構象,並以二維的Hi-C 矩陣作為預測輸出目標(圖1-2)。輸入訊息為 2 百萬鹼基對的 DNA,CTCF ChIP-seq 和 ATAC-seq。研究者使用 Onehot-encoding 來編碼離散的 DNA 序列,而 CTCF ChIP-seq 和 ATAC-seq 則編碼成非離散的特徵。

C.Origami 模型分為三個部分,處理並壓縮DNA 及基因組訊息的編碼器,Transformer 中間層和輸出Hi-C 解碼器#。其中編碼器由一系列 1D ResNet 和 strided convolution 構成用來編碼和壓縮 2 百萬鹼基對的輸入資訊。在編碼器末端 2 百萬長度的資訊被壓縮為 256 長度並作為 Transformer 的輸入資訊。 Transformer 的自註意力機制可以處理不同基因組區域間的 interdependency 並提升了模型的綜合性能。 Transformer 中的注意力矩陣還可以增強模型的可解釋性。研究者將注意力權重轉換成了 “attention score ”,用來衡量模型在預測時對於不同區域的重點。最後,研究者將 Transformer 模組的 1D 輸出用 “outer concatenation” 的方式轉換成了 2D 的 contact/adjacency matrix,用作 Hi-C 解碼器的輸入資訊。解碼器是一個 Dilated 2D ResNet。研究者們調整了不同層的 dilation factor 讓最後層的每個像素位置的 receptive field 都能覆蓋所有輸入資訊。
這個預測染色質構象的模型則稱為 C.Origami。研究者稱 C.Origami 是基因體學中第一個多模態深度學習模型。由於它多模態的特性,C.Origami 能夠準確地預測(de novo prediction)從未接觸過的新細胞類型的染色質構象。例如,在 IMR-90 細胞(肺成纖維細胞)上訓練的模型能夠準確預測 GM12878 細胞(B 淋巴細胞)裡特定的染色質構象(圖 3)。

圖3
#結構變異(structural variant)--- - 例如染色體易位---- 在腫瘤中非常常見,並經常改變染色質交互作用模式,進而可能影響癌基因或抑癌基因的表達。研究這些結構變異對染色質構象及基因表現的影響對理解腫瘤發生和進展的機制有重要作用。這類研究通常需要藉助 4C-seq 或 Hi-C 等實驗來分析結構變異位點的染色質構象,但往往受限於資源和時間的限制,難以大規模進行。
這項研究中,C.Origami 可以在輸入變數中模擬 DNA 序列的變異,然後預測變異後的癌症基因組中新的染色質交互作用。先前的研究發現 T 細胞急性淋巴性白血病(T-ALL)細胞模型 CUTLL1 有一個 chr7-chr9 的染色體轉位(圖 4)。透過計算模擬染色體易位變異,C.Origami 準確預測了在變異位點的新 TAD 結構,並檢測到從 chr9 延伸到 chr7 的『互動束』(chromatin stripe)結構(圖 4)。

#圖4
#鑑於C.Origami 的精確預測效果,並受到反向遺傳篩選原理的啟發,研究者提出了全新的高通量計算遺傳篩選(in silico genetic screening,ISGS) 方法,用以系統鑑定細胞類型特異性的功能基因組元件,並助力發現新的染色調控分子(圖5)。研究者基於 C.Origami 模型開發了用於系統性鑑定染色質構象所需的順式調控元件(cis-regulatory element)的計算遺傳篩選 ISGS 的框架。透過對全基因組 1kb 解析度的 ISGS,作者分離出對染色質構像有重要影響的順式調控元件(佔約 1% 的基因組)。這些染色質構象調控序列呈現對 CTCF 結合和 ATAC-seq 訊號的不同依賴(圖 5)。

#圖 5
#ISGS 框架可以對細胞或疾病特異性的染色質構象進行高通量篩選。研究者在 CUTLL1、Jurkat 和正常 T 細胞中分別進行了 ISGS,並發現了一個在 CHD4 基因附近的順式調控元件 (CHD4-insu) 在 T-ALL 細胞裡特異性缺失。篩選結果表明,T-ALL 細胞中 CHD4-insu 的絕緣性缺失可能使 CHD4 基因建立新的染色質相互作用,進而上調 CHD4 表達並促進白血病細胞增殖。
ISGS 也可以用來系統性地發現調控染色質構象的新型反式作用因子(trans-acting factors)。透過細胞類型特異性的重要調控序列與轉錄因子結合部位的富集分析,研究者確定了有助於細胞類型特異的基因組構象的調控因子。有趣的是,先前研究發現 MAZ 可能與 CTCF 一起調控染色質構象。透過 ISGS 及轉錄因子富集分析,作者發現 MAZ 大幅富集於開放染色質區域,而在 CTCF 結合的非開放染色質區域僅顯示微弱結合。這結果預示 MAZ 可能獨立於 CTCF 調節基因組構形。
研究者在染色質結構預測中看到了結合 DNA 序列與染色質資訊的多模態機器學習模型的巨大潛力。而此模型的底層多模態架構 Origami 可以推廣到其他基因體學資料的應用,例如表觀遺傳修飾、基因表現、突變功能性篩檢等。研究者預測,未來的基因組學研究將更多的轉向使用利用深度學習模型作為工具來進行主要計算遺傳篩選,並輔以生物實驗驗證的新一代高通量研究方法。
這項研究中,紐約大學醫學院博士研究生譚濟民為第一作者,Aristotelis Tsirigos 博士和夏波博士為共同通訊作者。這項研究起始於 2020 年 10 月疫情封控期間夏波與譚濟民的頭腦風暴,歷經兩年半的完善與打磨,在 2023 年一月於 Nature Biotechnology 正式刊出。
此專案的程式碼和訓練資料已在GitHub和Zenodo上開源,並配有Google Colab做功能展示。
計畫網址:https://github.com/tanjimin/C.Origami
##通訊作者夏波博士實驗室(Broad Institute of MIT and Harvard)首頁:www.boxialab.org
夏波博士致力於解析調控基因組三維構象的核心機制及其對人類疾病、發育及演化的生物學意義。夏波實驗室歡迎志同道合的博士後加入團隊。
Tsirigos Lab(New York University Grossman School of Medicine)首頁:http://www.tsirigos.com
Tsirigos Lab 的主要研究方向包括染色質,表觀遺傳學和機器學習在精準醫療上的應用。
以上是基因組構象預測模型與高通量計算遺傳篩選方法的探究與應用的詳細內容。更多資訊請關注PHP中文網其他相關文章!




