
蛋白质的动力学对于理解其机制至关重要。然而,通过计算预测蛋白质动学信息具有挑战性。
在此,来自山东大学、百图生科(BioMap)、北京理工大学、湖北医药学院、宁夏医科大学和阿卜杜拉国王科技大学(KAUST)的研究团队,提出了一个神经网络模型 RMSF-net,其优于以前的方法,并在大规模蛋白质动力学数据集中产生最佳结果;该模型可以在几秒钟内准确推断出蛋白质的动力学信息。
通过从实验蛋白质结构数据和低温电子显微镜 (cryo-EM) 数据集成中有效地学习,该方法能够准确识别低温电子显微镜图和 PDB 模型之间的交互式双向约束和监督,以最大限度地提高动力学预测效率。
RMSF-net 是一个可免费使用的工具,将在蛋白质动力学研究中发挥重要作用。
该研究以「Accurate Prediction of Protein Structural Flexibility by Deep Learning Integrating Intricate Atomic Structures and Cryo-EM Density Information」为题,于 7 月 2 日发布在《Nature Communications》上。

RMSF-net GitHub 地址:
蛋白质动力学
蛋白质动力学在理解其机制中至关重要。低温电子显微镜 (cryo-EM) 技术可解析大多数蛋白质,其中大分子结构由 3D 密度图表示。
低温电子显微镜的局限性
由于原始二维粒子图像的分辨率和信噪比较低,低温电子显微镜分析无法在重建过程中分辨出微小的构象变化。
深度学习在低温电子显微镜中的应用
深度学习方法广泛用于低温电子显微镜图的自动分析。利用高分辨率的低温电子显微镜图谱,可以从低温电子显微镜图谱中构建蛋白质数据库 (PDB) 模型。
RMSF-net 概述
RMSF-net 是一种神经网络模型,用于低温电子显微镜密度图。它利用低温电子显微镜密度和 PDB 模型信息,可以在几秒钟内准确推断蛋白质的动力学信息。
RMSF
RMSF 是一种广泛使用的测量方法,用于评估分子动力学 (MD) 分析中分子结构的灵活性。其主要目的是预测蛋白质内局部结构 (残基、原子) 的 RMSF。
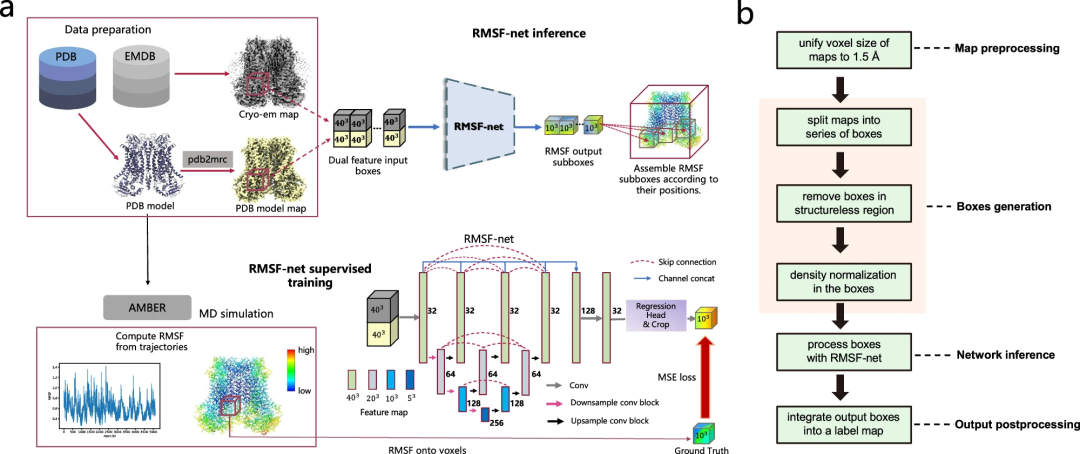
除了低温电子显微镜图之外,RMSF-net 还利用 PDB 模型作为额外输入,以产生非常接近 MD 模拟结果的 RMSF 预测。
RMSF-net 是一种三维卷积神经网络,包含两个相互连接的模块。主模块采用 Unet + (L3) 架构对输入密度框进行特征编码和解码。另一个模块利用 1x1 卷积对 Unet + 主干生成的特征图的通道进行回归。然后将中心裁剪应用于回归模块输出以获得中心 RMSF 子框,其中体素(voxel)值对应于其中包含的原子的 RMSF。最后,使用合并算法将 RMSF 子框在空间上合并为 RMSF 图。
此外,研究人员还构建了一个大规模蛋白质动力学数据集用于 RMSF-net 的训练和验证,其中选择了 335 个具有拟合 PDB 模型的低温电子显微镜结构条目并执行相应的 MD 模拟。综合实验结果证明了 RMSF-net 的效率和有效性。
表:不同 RMSF 预测方法在数据集上的表现。(来源:论文)
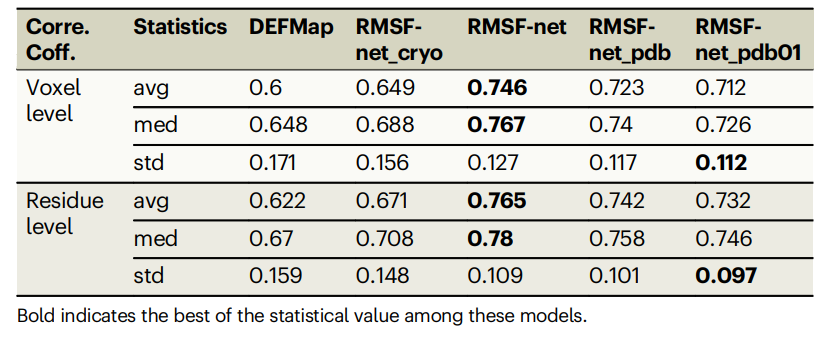
RMSF-net 在严格的 5 倍交叉验证中表现出色,与 MD 模拟结果的相关系数达到 0.746±0.127。与 DEFMap 相比,RMSF-net 的相关系数提高了 15%,与基线方法相比,提高了 10%。
动力学预测的可解释性
研究人员通过对比实验增强了 RMSF-net 动力学预测的可解释性。他们将 RMSF 预测过程分为两个步骤:
研究表明,基于低温电子显微镜图谱的模型(例如 DEFMap 或 RMSF-net_cryo)的动力学预测主要通过解读蛋白质结构实现。这突出了蛋白质拓扑结构与动力学之间的联系,符合结构-功能关系的第一原理。
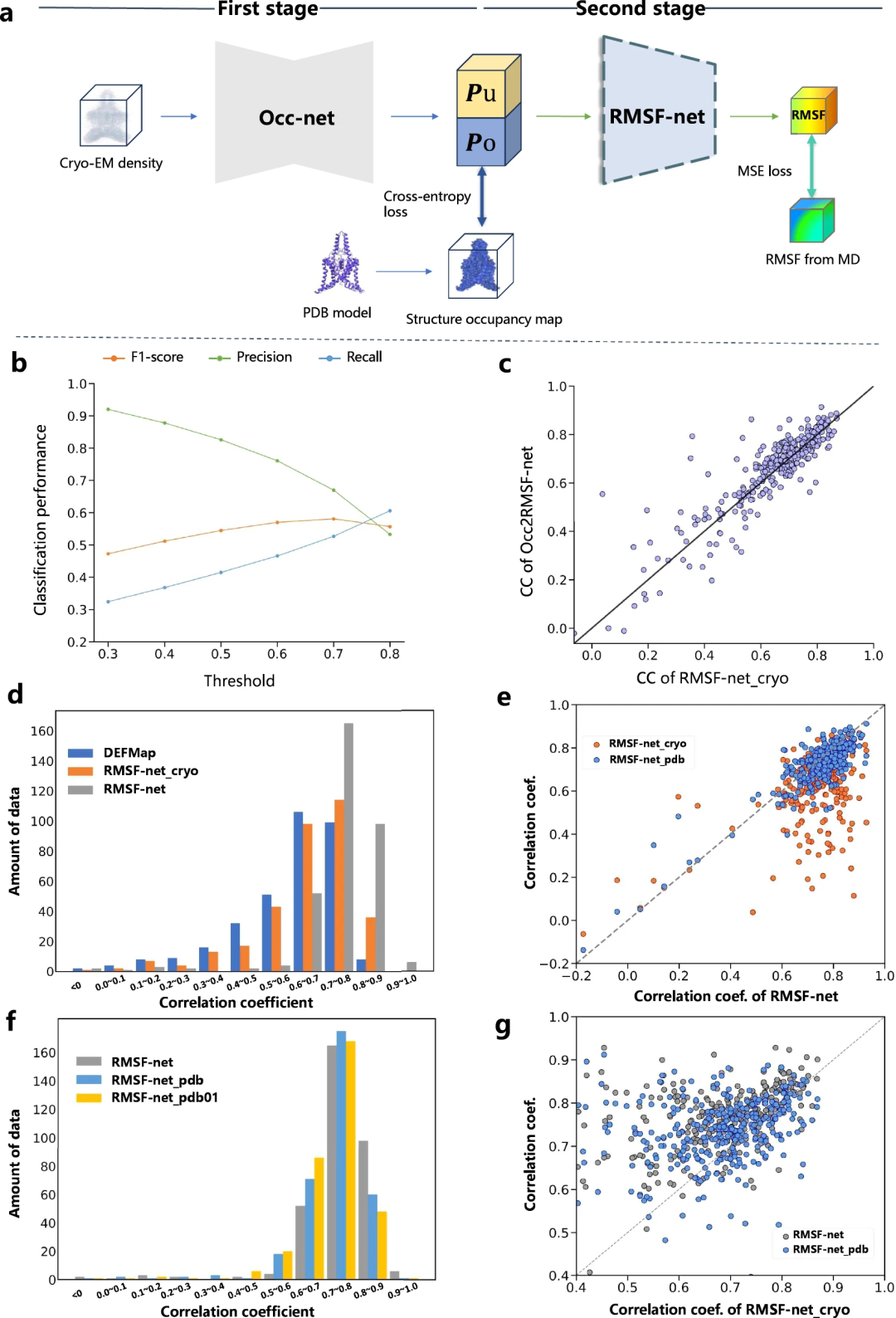
此外,通过对 RMSF-net_cryo、RMSF-net_pdb 和最终的双组合 RMSF-net 进行全面比较,证明了:一方面,来自 PDB 模型的结构信息在 RMSF-net 中起主要作用,其中深度模型从 MD 模拟中学习结构拓扑和灵活性之间的模式,另一方面,低温电子显微镜图谱异质密度分布中包含的动力学信息进一步增强了模型。这些结果验证了低温电子显微镜图和 PDB 模型的信息对 RMSF-net 中的蛋白质动力学预测的互补作用。
局限性与未来方向
不可否认的是,RMSF-net 主要限于预测纯蛋白质及其复合物在溶液中的柔韧性。对于蛋白质在与小分子配体结合或在膜环境中的动力学特性,该方法在某些局部区域可能会表现出不准确性。
RMSF-net 的卓越性能揭示了进一步研究该方向的可行性。该研究还没有扩展到核酸和蛋白质-核酸复合物。综合表征大分子动力学的各个方面,包括多构象预测和转变分析,在未来需要进一步进行广泛而深入的研究。
尽管如此,作为预测蛋白质动力学的工具,RMSF-net 由于其优越的性能和超快的处理速度,在蛋白质结构和动力学研究中仍有很大的应用前景。
注:封面来自网络
以上是仅几秒,准确推断蛋白动力学信息,山大、北理工等AI模型RMSF-net登Nature子刊的详细内容。更多信息请关注PHP中文网其他相关文章!




