提速1200倍!MIT开发新一代药物研发AI,吊打老模型
众所周知,整个宇宙充满着无数分子。
这些分子中又有多少具有潜在的类似药物的特性,可用于开发挽救生命的药物呢?是百万级?还是十亿级?又或是万亿级?
答案是:10的60次幂。
如此巨大的数字,大大延缓了新药的研发进度,像新冠等快速传播的疾病,目前迟迟未有特效药,也是因为分子种类和数量太大,远远超出了现有药物设计模型可以计算的范围。
MIT的一个研究团队偏不信这个邪。算不过来是吧,那把之前的模型加加速总可以吧?
这一加速,就是1200倍。
他们研究了一款名为「EquiBind」的几何深度学习模型,该模型比之前最快的计算分子对接模型「QuickVina2-W」快了1200倍, 成功地将类药物分子与蛋白质结合,减少了药物试验失败的机会和成本。
研究论文即将发表在ICML 2022上。
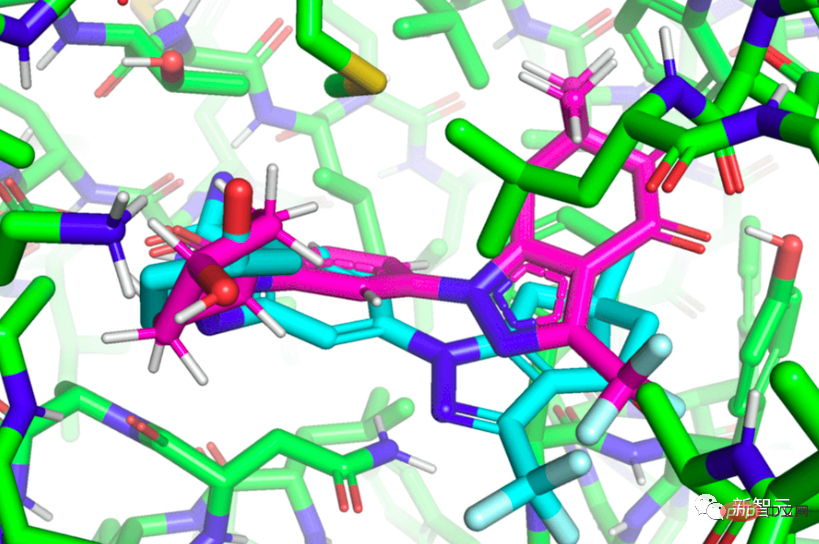
初识「EquiBind」
「EquiBind」是基于其前身「EquiDock」研发的,「EquiDock」使用了已故的麻省理工学院AI研究员Octavian-Eugen Ganea开发的技术,去结合两种蛋白质。Ganea也是「EquiBind」论文的合著者。
在药物开发开始之前,研究人员必须找到有前途的药物样分子,这些分子可以在药物发现的过程中正确地结合或「停靠」在特定的蛋白质靶点上。
在与蛋白质成功对接后,结合药物(配体),可以阻止蛋白质发挥作用。如果这种情况发生在细菌的一种基本蛋白质上,它可以杀死细菌,从而保护人体。
然而,不论是从经济角度分析,还是从计算角度分析,药物发现的过程都可能是昂贵的,研发过程动辄耗资数十亿美元,并且在FDA最终批准之前,会进行超过十年的开发和测试。
更重要的是,90%的药物在人体试验后由于没有效果或副作用过多而失败。
所以制药公司收回这些成本的方法之一就是,提高最终成功研发药物的价格。
走进「EquiBind」
目前,寻找有希望的候选药物分子的计算过程是这样的:大多数最先进的计算模型依赖于大量的候选样本,再加上评分、排名和微调等方法,以获得配体和蛋白质之间的最佳「匹配」。
Hannes Stärk是MIT电子工程与计算机科学系的一年级研究生,也是这篇论文的主要作者,他把典型的「配体-蛋白质」结合方法比作「试图把钥匙放进有很多钥匙孔的锁中」。

典型的模型在选择最佳模型之前会花费时间对每个「拟合度」进行评分。相比之下,「EquiBind」无需事先了解蛋白质的目标口袋,只需一步就能直接预测出精确的关键位置,这被称为「盲对接」。
与大多数需要多次尝试才能找到配体在蛋白质中的有利位置的模型不同,「EquiBind」已经具有内置的几何推理功能,可帮助模型学习分子的潜在物理特性,并成功地进行归纳。以便在遇到新的或者无法识别的数据时成功泛化,以做出更好的预测。
这些发现的发布迅速引起了业内专业人士的注意,其中包括Relay Therapeutics首席数据官Pat Walters。
Walters建议,研究小组可以在一种现有的用于肺癌、白血病和胃肠道肿瘤的药物和蛋白质上去尝试他们的模型。尽管大多数传统的对接方法都未能成功地结合这些蛋白质上的配体,但是「EquiBind」成功了。
Walters说:「EquiBind为对接问题提供了一种独特的解决方案,它结合了姿态预测和结合位点识别。」
「并且这种方法利用了来自数千个公开的晶体结构的信息,有可能会以新的方式影响该领域。”
Stärk表示:「当其他所有方法都完全错误或仅有一个正确时,我们很惊讶,因为EquiBind能够把它放进正确的口袋,看到这个结果,我们是十分激动的!」
助力「EquiBind」
尽管「EquiBind」已经收到了大量的来自业内专业人士的反馈,并且这些反馈帮助了团队考虑计算模型的实际用途,但Stärk仍希望在7月份即将举行的ICML上找到不同的观点。
Stärk表示:「我最期待的反馈是关于如何进一步改进该模型的建议。」
「我想和这些研究人员讨论,告诉他们我认为下一步可以怎么做,并鼓励他们继续前进,将模型用于他们自己的论文和方法中。目前已经有很多研究人员联系了我们,询问我们这个模型是否会对他们的问题有用。」
此外,这篇文章同样也是为了纪念Octavian-Eugen Gane,他为几何机器学习研究做出了至关重要的贡献,并慷慨地指导了许多学生,他是一个有着谦逊灵魂的杰出学者。
今年上半年,他在一次徒步旅行中永远离开了我们。
以上是提速1200倍!MIT开发新一代药物研发AI,吊打老模型的详细内容。更多信息请关注PHP中文网其他相关文章!

热AI工具

Undresser.AI Undress
人工智能驱动的应用程序,用于创建逼真的裸体照片

AI Clothes Remover
用于从照片中去除衣服的在线人工智能工具。

Undress AI Tool
免费脱衣服图片

Clothoff.io
AI脱衣机

AI Hentai Generator
免费生成ai无尽的。

热门文章

热工具

记事本++7.3.1
好用且免费的代码编辑器

SublimeText3汉化版
中文版,非常好用

禅工作室 13.0.1
功能强大的PHP集成开发环境

Dreamweaver CS6
视觉化网页开发工具

SublimeText3 Mac版
神级代码编辑软件(SublimeText3)

热门话题
 全球最强开源 MoE 模型来了,中文能力比肩 GPT-4,价格仅为 GPT-4-Turbo 的近百分之一
May 07, 2024 pm 04:13 PM
全球最强开源 MoE 模型来了,中文能力比肩 GPT-4,价格仅为 GPT-4-Turbo 的近百分之一
May 07, 2024 pm 04:13 PM
想象一下,一个人工智能模型,不仅拥有超越传统计算的能力,还能以更低的成本实现更高效的性能。这不是科幻,DeepSeek-V2[1],全球最强开源MoE模型来了。DeepSeek-V2是一个强大的专家混合(MoE)语言模型,具有训练经济、推理高效的特点。它由236B个参数组成,其中21B个参数用于激活每个标记。与DeepSeek67B相比,DeepSeek-V2性能更强,同时节省了42.5%的训练成本,减少了93.3%的KV缓存,最大生成吞吐量提高到5.76倍。DeepSeek是一家探索通用人工智
 AI颠覆数学研究!菲尔兹奖得主、华裔数学家领衔11篇顶刊论文|陶哲轩转赞
Apr 09, 2024 am 11:52 AM
AI颠覆数学研究!菲尔兹奖得主、华裔数学家领衔11篇顶刊论文|陶哲轩转赞
Apr 09, 2024 am 11:52 AM
AI,的确正在改变数学。最近,一直十分关注这个议题的陶哲轩,转发了最近一期的《美国数学学会通报》(BulletinoftheAmericanMathematicalSociety)。围绕「机器会改变数学吗?」这个话题,众多数学家发表了自己的观点,全程火花四射,内容硬核,精彩纷呈。作者阵容强大,包括菲尔兹奖得主AkshayVenkatesh、华裔数学家郑乐隽、纽大计算机科学家ErnestDavis等多位业界知名学者。AI的世界已经发生了天翻地覆的变化,要知道,其中很多文章是在一年前提交的,而在这一
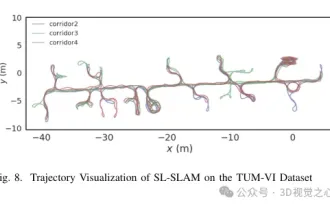 超越ORB-SLAM3!SL-SLAM:低光、严重抖动和弱纹理场景全搞定
May 30, 2024 am 09:35 AM
超越ORB-SLAM3!SL-SLAM:低光、严重抖动和弱纹理场景全搞定
May 30, 2024 am 09:35 AM
写在前面今天我们探讨下深度学习技术如何改善在复杂环境中基于视觉的SLAM(同时定位与地图构建)性能。通过将深度特征提取和深度匹配方法相结合,这里介绍了一种多功能的混合视觉SLAM系统,旨在提高在诸如低光条件、动态光照、弱纹理区域和严重抖动等挑战性场景中的适应性。我们的系统支持多种模式,包括拓展单目、立体、单目-惯性以及立体-惯性配置。除此之外,还分析了如何将视觉SLAM与深度学习方法相结合,以启发其他研究。通过在公共数据集和自采样数据上的广泛实验,展示了SL-SLAM在定位精度和跟踪鲁棒性方面优
 你好,电动Atlas!波士顿动力机器人复活,180度诡异动作吓坏马斯克
Apr 18, 2024 pm 07:58 PM
你好,电动Atlas!波士顿动力机器人复活,180度诡异动作吓坏马斯克
Apr 18, 2024 pm 07:58 PM
波士顿动力Atlas,正式进入电动机器人时代!昨天,液压Atlas刚刚「含泪」退出历史舞台,今天波士顿动力就宣布:电动Atlas上岗。看来,在商用人形机器人领域,波士顿动力是下定决心要和特斯拉硬刚一把了。新视频放出后,短短十几小时内,就已经有一百多万观看。旧人离去,新角色登场,这是历史的必然。毫无疑问,今年是人形机器人的爆发年。网友锐评:机器人的进步,让今年看起来像人类的开幕式动作、自由度远超人类,但这真不是恐怖片?视频一开始,Atlas平静地躺在地上,看起来应该是仰面朝天。接下来,让人惊掉下巴
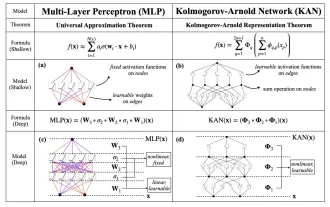 替代MLP的KAN,被开源项目扩展到卷积了
Jun 01, 2024 pm 10:03 PM
替代MLP的KAN,被开源项目扩展到卷积了
Jun 01, 2024 pm 10:03 PM
本月初,来自MIT等机构的研究者提出了一种非常有潜力的MLP替代方法——KAN。KAN在准确性和可解释性方面表现优于MLP。而且它能以非常少的参数量胜过以更大参数量运行的MLP。比如,作者表示,他们用KAN以更小的网络和更高的自动化程度重现了DeepMind的结果。具体来说,DeepMind的MLP有大约300,000个参数,而KAN只有约200个参数。KAN与MLP一样具有强大的数学基础,MLP基于通用逼近定理,而KAN基于Kolmogorov-Arnold表示定理。如下图所示,KAN在边上具
 谷歌狂喜:JAX性能超越Pytorch、TensorFlow!或成GPU推理训练最快选择
Apr 01, 2024 pm 07:46 PM
谷歌狂喜:JAX性能超越Pytorch、TensorFlow!或成GPU推理训练最快选择
Apr 01, 2024 pm 07:46 PM
谷歌力推的JAX在最近的基准测试中性能已经超过Pytorch和TensorFlow,7项指标排名第一。而且测试并不是在JAX性能表现最好的TPU上完成的。虽然现在在开发者中,Pytorch依然比Tensorflow更受欢迎。但未来,也许有更多的大模型会基于JAX平台进行训练和运行。模型最近,Keras团队为三个后端(TensorFlow、JAX、PyTorch)与原生PyTorch实现以及搭配TensorFlow的Keras2进行了基准测试。首先,他们为生成式和非生成式人工智能任务选择了一组主流
 特斯拉机器人进厂打工,马斯克:手的自由度今年将达到22个!
May 06, 2024 pm 04:13 PM
特斯拉机器人进厂打工,马斯克:手的自由度今年将达到22个!
May 06, 2024 pm 04:13 PM
特斯拉机器人Optimus最新视频出炉,已经可以在厂子里打工了。正常速度下,它分拣电池(特斯拉的4680电池)是这样的:官方还放出了20倍速下的样子——在小小的“工位”上,拣啊拣啊拣:这次放出的视频亮点之一在于Optimus在厂子里完成这项工作,是完全自主的,全程没有人为的干预。并且在Optimus的视角之下,它还可以把放歪了的电池重新捡起来放置,主打一个自动纠错:对于Optimus的手,英伟达科学家JimFan给出了高度的评价:Optimus的手是全球五指机器人里最灵巧的之一。它的手不仅有触觉
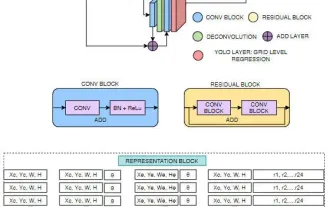 FisheyeDetNet:首个基于鱼眼相机的目标检测算法
Apr 26, 2024 am 11:37 AM
FisheyeDetNet:首个基于鱼眼相机的目标检测算法
Apr 26, 2024 am 11:37 AM
目标检测在自动驾驶系统当中是一个比较成熟的问题,其中行人检测是最早得以部署算法之一。在多数论文当中已经进行了非常全面的研究。然而,利用鱼眼相机进行环视的距离感知相对来说研究较少。由于径向畸变大,标准的边界框表示在鱼眼相机当中很难实施。为了缓解上述描述,我们探索了扩展边界框、椭圆、通用多边形设计为极坐标/角度表示,并定义一个实例分割mIOU度量来分析这些表示。所提出的具有多边形形状的模型fisheyeDetNet优于其他模型,并同时在用于自动驾驶的Valeo鱼眼相机数据集上实现了49.5%的mAP






