Meta AI开放6亿+宏基因组蛋白质结构图谱,150亿语言模型用两周完成
今年,DeepMind 公布了大约 2.2 亿种蛋白质的预测结构,它几乎涵盖了 DNA 数据库中已知生物体的所有蛋白质。现在,另一家科技巨头 Meta 正在填补另一空白,微生物领域。
简单来说,Meta 使用 AI 技术预测了约 6 亿种蛋白质结构,这些蛋白质来自细菌和其他尚未被表征的微生物。团队负责人 Alexander Rives 表示:「这些蛋白质是我们所知最少的结构,它们是非常神秘的蛋白质。我认为这些发现为深入了解生物学提供了潜力。」
通常,语言模型是在大量文本上进行训练的。Meta 为了将语言模型应用于蛋白质,Rives 及其同事将已知的蛋白质序列作为输入,这些蛋白质由 20 种氨基酸组成,并用不同的字母表示。然后,该网络在遮蔽一定比例氨基酸的情况下学会了自动补全蛋白质。
Meta 将这个网络命名为 ESMFold。虽然 ESMFold 预测准确性不如 AlphaFold,但在预测结构方面,它比 AlphaFold 快约 60 倍。这一速度意味着可以将蛋白质结构预测扩展到更大的数据库。

- 论文地址:https://www.biorxiv.org/content/10.1101/2022.07.20.500902v2
- 项目地址:https://github.com/facebookresearch/esm
如今,作为测试,Meta 决定将他们的模型应用于宏基因组 DNA 数据库,这些 DNA 全部来自环境,包括土壤、海水、人类肠道、皮肤和其他微生物栖息地。Meta AI 宣布推出包含 6 亿多个蛋白质的 ESM 宏基因组图谱(ESM Metagenomic Atlas),它是首个蛋白质宇宙「暗物质」的综合视图。这还是最大的高分辨率预测结构数据库,比任何现有的蛋白质结构数据库都要大 3 倍,并且是第一个全面、大规模地涵盖宏基因组蛋白质的数据库。
Meta 团队总共预测了超过 6.17 亿个蛋白质结构,只花了两周的时间。Rives 说,预测是免费的,任何人都可以使用,就像模型的底层代码一样。
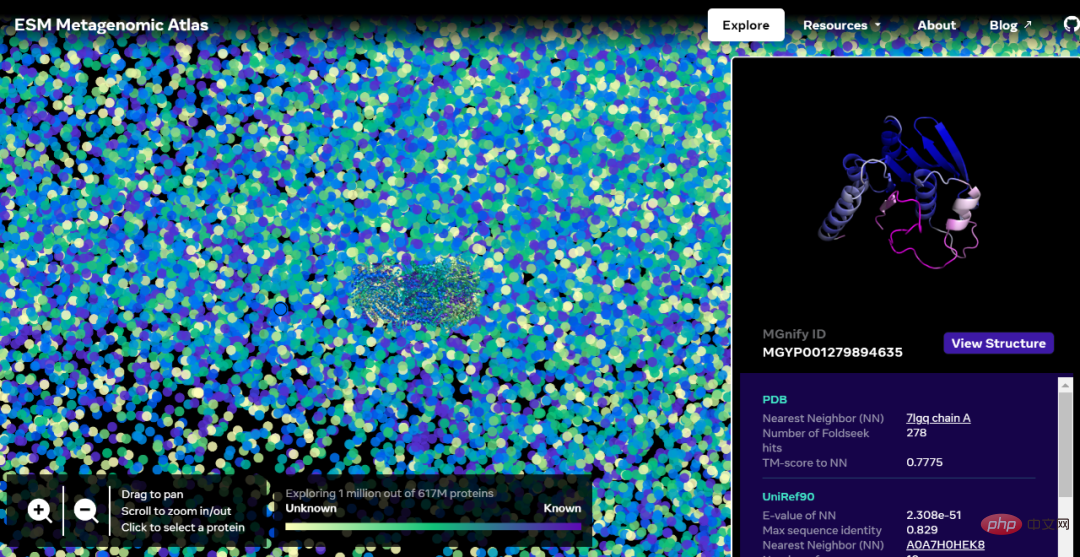
交互版本地址:https://esmatlas.com/explore?at=1%2C1%2C21.999999344348925
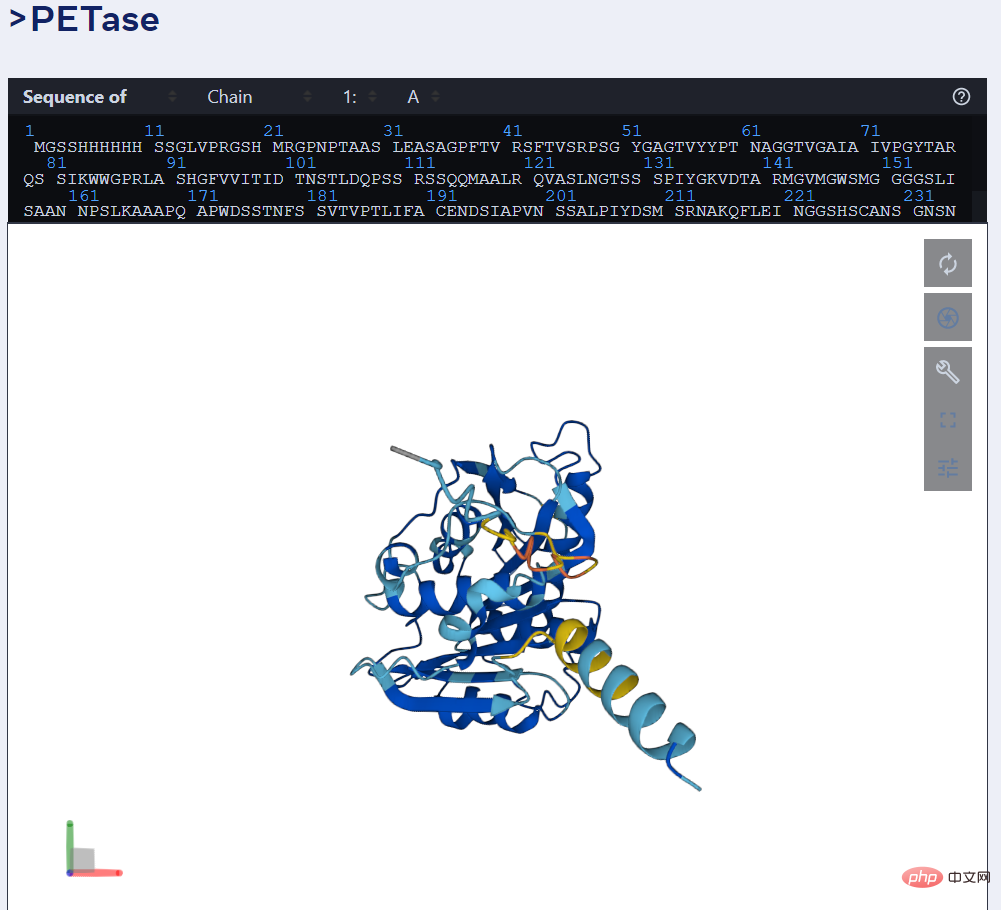
举例而言,下图为 ESMFold 对 PET 酶的预测。
引言
众所周知,蛋白质作为复杂且动态的分子,其由基因编码,主要负责生命基本过程。蛋白质在生物学中有着惊人作用。比如,人类眼睛中的视杆和视锥细胞可以感知光线,因而我们能看到外面的世界;构成听觉和触觉基础的分子传感器;植物中把光能转化为化学能的复杂分子;驱动微生物和人类肌肉运动的「马达」;分解塑料的酶;保护我们免受疾病的抗体,等等这些都是蛋白质。
1998 年,来自威斯康辛大学植物病理学部门的 Jo Handelsman 首次提出宏基因组学(Metagenomics)这一概念,它是源于将来自环境中基因集可以在某种程度上当成单个基因组研究分析的想法,而宏的英文正是 meta-,也翻译为元。
宏基因组学揭示了数十亿个对科学来说是新的蛋白质序列,并首次编入由 NCBI、欧洲生物信息学研究所 (European Bioinformatics Institute) 和联合基因组研究所 (Joint Genome Institute) 等公共项目编制的大型数据库中。
Meta AI 开发的新的蛋白质折叠方法,该方法利用大型语言模型,在宏基因组数据库中(具有数亿蛋白质)创建了首个全面的蛋白质结构视图。Meta 发现,相对于现有的 SOTA 蛋白质结构预测方法,语言模型可以将预测蛋白质原子级三维结构的速度提高 60 倍。这一进展将有助于加速蛋白质结构理解的新时代,这是首次人类有可能了解基因测序技术正在编目的数十亿蛋白质的结构。
解锁隐藏的自然世界:宏基因组结构空间的首个综合视图
我们知道,基因测序的进步使得对数十亿个宏基因组蛋白序列进行编目成为可能。但是,通过实验确定数以亿计蛋白质的 3D 结构远远超出了时间密集型实验室技术的范围,例如 X 射线晶体学,它可能需要数周乃至数年的时间来检测单个蛋白质。计算方式可以让我们深入了解使用实验技术无法实现的宏基因组学蛋白质。
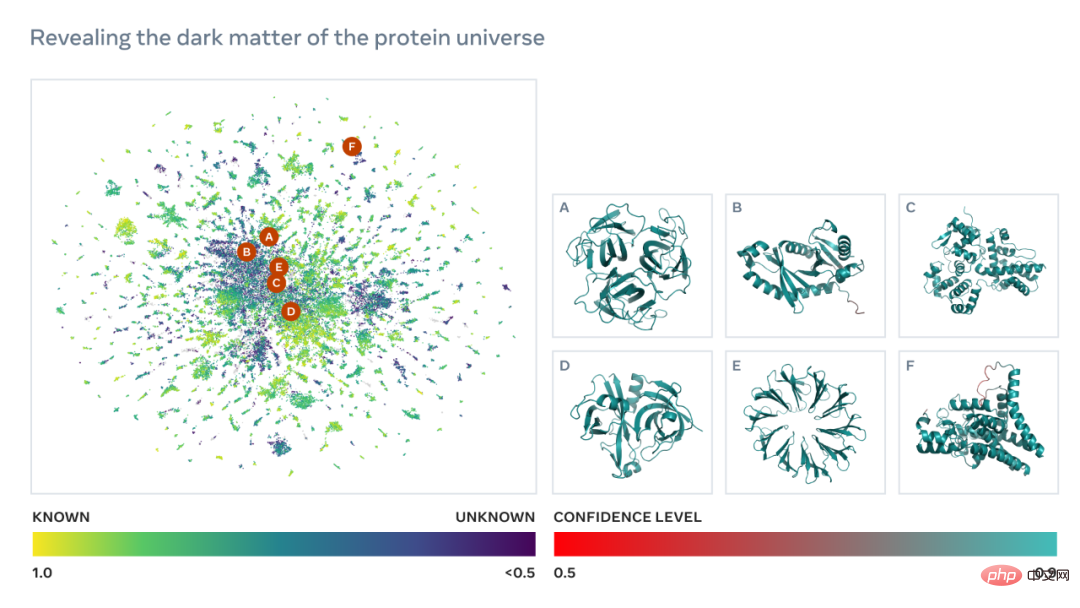
ESM 宏基因组图谱将使科学家能够在数亿蛋白质的尺度上搜索和分析宏基因组蛋白质的结构。这可以帮助识别以前未被表征的结构,寻找遥远的进化关系,并发现可用于医学和其他应用的新蛋白质。
如下为一张包含数以万计高置信度预测的图谱,展示了与目前已知结构的蛋白质的相似性。并且,该图像首次显示了完全未知的蛋白质结构空间的更大区域。
学习阅读生物学语言
如下图所示,ESM-2 语言模型经过训练,可以预测进化过程中被序列掩盖的氨基酸。Meta AI 发现,作为训练的结果,蛋白质结构的信息出现在该模型的内部状态中。这实在令人惊讶,因为该模型仅在序列上进行了训练。
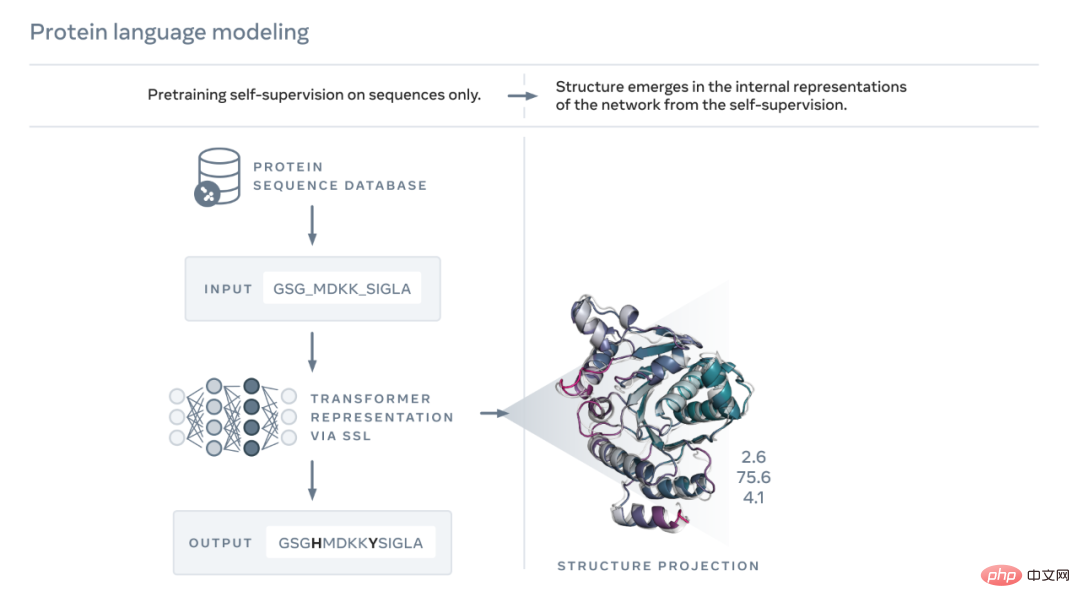
就像论文或信件的文本一样,蛋白质可以写成字符序列。其中,每个字符对应 20 种标准化学元素(氨基酸)中的一种,每种又具有不同的特性,它们是蛋白质的构建块。这些构建块能够以天文数字的不同方式组合在一起,例如对于由 200 个氨基酸组成的蛋白质,存在 20^200 个可能的序列,这要比可见宇宙中的原子数量还要多。每个序列都折叠成 3D 形状(但并非所有序列都会折叠成连贯的结构,许多序列折叠成无序形式),正是这种形状在很大程度上决定了蛋白质的生物学功能。
学习阅读这种生物学语言带来了很大挑战。虽然蛋白质序列和文本段落都可以写成字符,但它们之间存在着深刻而根本性的差异。蛋白质序列描述了一个分子的化学结构,该分子根据物理定律折叠成复杂的 3D 形状。
蛋白质序列包含了传递蛋白质折叠结构信息的统计模式。举例而言,如果一个蛋白质中的两个位置共同进化,或者换言之,如果其中一个位置出现某种氨基酸,通常与另一个位置的某种氨基酸配对,这可能意味着这两个位置在折叠结构中相互作用。这类似于拼图游戏中的两块拼图,进化必须选择在折叠结构中拼合在一起的氨基酸。这又意味着我们通常可以通过观察蛋白质序列中的模式来推断蛋白质的结构。
ESM 使用 AI 来学习阅读这些模式。2019 年,Meta AI 提供证据证明语言模型学习了蛋白质的特性,例如它们的结构和功能。通过一种被称为掩码语言建模的自我监督学习形式,Meta AI 在数百万个天然蛋白质的序列上训练了一个语言模型。使用这种方法,模型必须正确填写文本段落中的空白,例如「To _ or not to , that is the _____」。
之后,Meta AI 训练了一个语言模型来填补蛋白质序列中的空白。他们发现,蛋白质结构和功能的信息在这一训练中浮现了出来。2020 年,Meta 发布了一个 SOTA 蛋白质语言模型 ESM1b,用于各种应用,包括帮助科学家预测 COVID-19 的演变以及发现疾病的遗传原因。
现在,Meta AI 扩展了这种方法,用来创建下一代蛋白质语言模型 ESM-2,它的参数为 150 亿,是迄今为止最大的蛋白质语言模型。他们发现,当模型参数从 800 万放大到 150 亿时,内部表示中会出现信息,从而能够以原子分辨率进行 3D 结构预测。
将蛋白质折叠实现数量级加速
在下图中,随着模型的扩大,高分辨率的蛋白质结构出现。同时随着模型的缩放,蛋白质结构的原子分辨率图像中会出现新的细节。

使用当前 SOTA 计算工具,在实际时间范围内预测数亿蛋白质序列结构可能花费数年时间,即便用上主要研究机构的资源也是如此。因此,想要在宏基因组尺度上进行预测,预测速度的突破至关重要。
Meta AI 发现使用蛋白质序列的语言模型大大加快了结构预测的速度,最高提升 60 倍。这足以在短短几周内对整个宏基因组数据库做出预测,并且可以扩展到比我们当前发布的数据库大得多的数据库。事实上,这种新的结构预测能力能够在短短两周内,在大约 2000 个 GPU 组成的集群上预测超过 6 亿多个宏基因组蛋白的序列。
此外,当前 SOTA 结构预测方法需要搜索大型蛋白质数据库以识别相关序列。这些方法实际上需要一整组进化相关的序列作为输入,以便它们可以提取与结构相关的模式。Meta AI 的 ESM-2 语言模型在其对蛋白质序列的训练过程中学习这些进化模式,进而能够直接从蛋白质序列中对 3D 结构进行高分辨率预测。
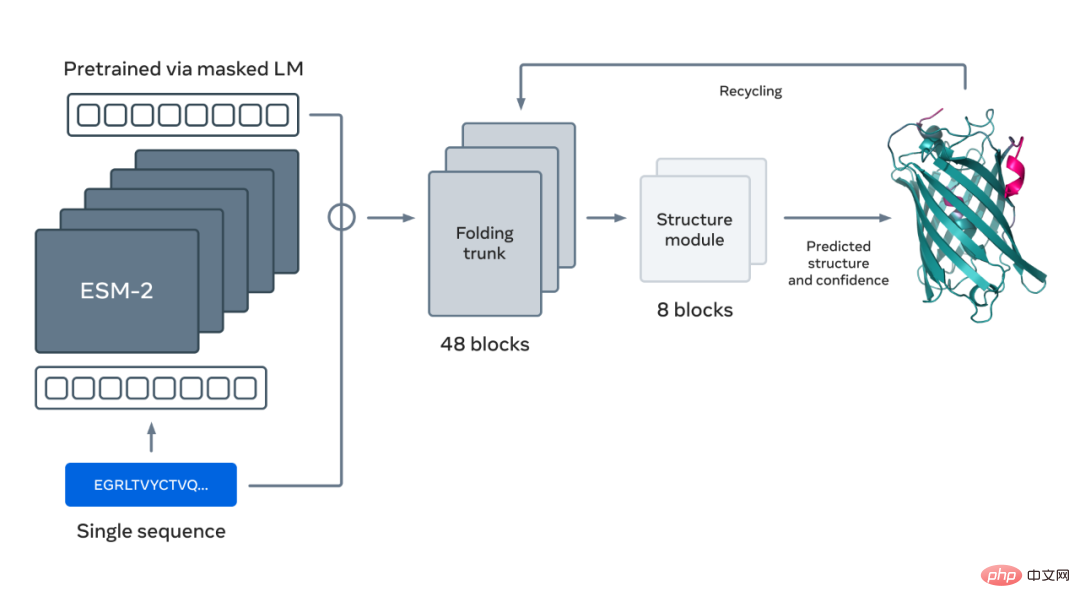
下图展示了使用 ESM-2 语言模型进行蛋白质折叠。箭头从左到右显示了网络中从语言模型到折叠 trunk 再到结构模块的信息流,最后输出 3D 坐标和置信度。
更多详细内容请参阅原文。
博客链接:https://ai.facebook.com/blog/protein-folding-esmfold-metagenomics/
以上是Meta AI开放6亿+宏基因组蛋白质结构图谱,150亿语言模型用两周完成的详细内容。更多信息请关注PHP中文网其他相关文章!

热AI工具

Undresser.AI Undress
人工智能驱动的应用程序,用于创建逼真的裸体照片

AI Clothes Remover
用于从照片中去除衣服的在线人工智能工具。

Undress AI Tool
免费脱衣服图片

Clothoff.io
AI脱衣机

AI Hentai Generator
免费生成ai无尽的。

热门文章

热工具

记事本++7.3.1
好用且免费的代码编辑器

SublimeText3汉化版
中文版,非常好用

禅工作室 13.0.1
功能强大的PHP集成开发环境

Dreamweaver CS6
视觉化网页开发工具

SublimeText3 Mac版
神级代码编辑软件(SublimeText3)

热门话题
 如何检查CentOS HDFS配置
Apr 14, 2025 pm 07:21 PM
如何检查CentOS HDFS配置
Apr 14, 2025 pm 07:21 PM
检查CentOS系统中HDFS配置的完整指南本文将指导您如何有效地检查CentOS系统上HDFS的配置和运行状态。以下步骤将帮助您全面了解HDFS的设置和运行情况。验证Hadoop环境变量:首先,确认Hadoop环境变量已正确设置。在终端执行以下命令,验证Hadoop是否已正确安装并配置:hadoopversion检查HDFS配置文件:HDFS的核心配置文件位于/etc/hadoop/conf/目录下,其中core-site.xml和hdfs-site.xml至关重要。使用
 centos关机命令行
Apr 14, 2025 pm 09:12 PM
centos关机命令行
Apr 14, 2025 pm 09:12 PM
CentOS 关机命令为 shutdown,语法为 shutdown [选项] 时间 [信息]。选项包括:-h 立即停止系统;-P 关机后关电源;-r 重新启动;-t 等待时间。时间可指定为立即 (now)、分钟数 ( minutes) 或特定时间 (hh:mm)。可添加信息在系统消息中显示。
 CentOS上GitLab的备份方法有哪些
Apr 14, 2025 pm 05:33 PM
CentOS上GitLab的备份方法有哪些
Apr 14, 2025 pm 05:33 PM
CentOS系统下GitLab的备份与恢复策略为了保障数据安全和可恢复性,CentOS上的GitLab提供了多种备份方法。本文将详细介绍几种常见的备份方法、配置参数以及恢复流程,帮助您建立完善的GitLab备份与恢复策略。一、手动备份利用gitlab-rakegitlab:backup:create命令即可执行手动备份。此命令会备份GitLab仓库、数据库、用户、用户组、密钥和权限等关键信息。默认备份文件存储于/var/opt/gitlab/backups目录,您可通过修改/etc/gitlab
 centos安装mysql
Apr 14, 2025 pm 08:09 PM
centos安装mysql
Apr 14, 2025 pm 08:09 PM
在 CentOS 上安装 MySQL 涉及以下步骤:添加合适的 MySQL yum 源。执行 yum install mysql-server 命令以安装 MySQL 服务器。使用 mysql_secure_installation 命令进行安全设置,例如设置 root 用户密码。根据需要自定义 MySQL 配置文件。调整 MySQL 参数和优化数据库以提升性能。
 CentOS上PyTorch的分布式训练如何操作
Apr 14, 2025 pm 06:36 PM
CentOS上PyTorch的分布式训练如何操作
Apr 14, 2025 pm 06:36 PM
在CentOS系统上进行PyTorch分布式训练,需要按照以下步骤操作:PyTorch安装:前提是CentOS系统已安装Python和pip。根据您的CUDA版本,从PyTorch官网获取合适的安装命令。对于仅需CPU的训练,可以使用以下命令:pipinstalltorchtorchvisiontorchaudio如需GPU支持,请确保已安装对应版本的CUDA和cuDNN,并使用相应的PyTorch版本进行安装。分布式环境配置:分布式训练通常需要多台机器或单机多GPU。所
 docker原理详解
Apr 14, 2025 pm 11:57 PM
docker原理详解
Apr 14, 2025 pm 11:57 PM
Docker利用Linux内核特性,提供高效、隔离的应用运行环境。其工作原理如下:1. 镜像作为只读模板,包含运行应用所需的一切;2. 联合文件系统(UnionFS)层叠多个文件系统,只存储差异部分,节省空间并加快速度;3. 守护进程管理镜像和容器,客户端用于交互;4. Namespaces和cgroups实现容器隔离和资源限制;5. 多种网络模式支持容器互联。理解这些核心概念,才能更好地利用Docker。
 CentOS下GitLab的日志如何查看
Apr 14, 2025 pm 06:18 PM
CentOS下GitLab的日志如何查看
Apr 14, 2025 pm 06:18 PM
CentOS系统下查看GitLab日志的完整指南本文将指导您如何查看CentOS系统中GitLab的各种日志,包括主要日志、异常日志以及其他相关日志。请注意,日志文件路径可能因GitLab版本和安装方式而异,若以下路径不存在,请检查GitLab安装目录及配置文件。一、查看GitLab主要日志使用以下命令查看GitLabRails应用程序的主要日志文件:命令:sudocat/var/log/gitlab/gitlab-rails/production.log此命令会显示produc
 CentOS上PyTorch的GPU支持情况如何
Apr 14, 2025 pm 06:48 PM
CentOS上PyTorch的GPU支持情况如何
Apr 14, 2025 pm 06:48 PM
在CentOS系统上启用PyTorchGPU加速,需要安装CUDA、cuDNN以及PyTorch的GPU版本。以下步骤将引导您完成这一过程:CUDA和cuDNN安装确定CUDA版本兼容性:使用nvidia-smi命令查看您的NVIDIA显卡支持的CUDA版本。例如,您的MX450显卡可能支持CUDA11.1或更高版本。下载并安装CUDAToolkit:访问NVIDIACUDAToolkit官网,根据您显卡支持的最高CUDA版本下载并安装相应的版本。安装cuDNN库:前






