华人团队成功开发AI预测癌症患者适用药物,成果发表在Nature子刊上
只需一个AI,9808名癌症患者对药物的临床反应,全能预测。
而且结果和临床观察表现一致。
这就是由纽约市立大学Lei Xie团队带来的最新成果CODE-AE(context-aware deconfounding autoencoder)。

它提出一种新型的上下文自编码模型,可以预测不同患者对药物的特异性反应。
这将对新药开发和临床试验产生重大影响。
要知道,传统模式下一种新药开发、试验、完全上市,中间需要近10年的时间,消耗的资金也空前庞大,动辄就是10亿美元。
周期会如此之长,是因为新药在人体内的反应难以预测,往往需要反复试验进行测试。
而如果AI能够利用数据进行预测,将大幅缩短新药上市时间,降低成本。
目前,该研究登上Nature子刊《Nature Machine Intelligence》。
简单来说,CODE-AE是利用新药在体外细胞验证上的数据,来预测药物在人体身上会产生的反应。
这样就避免了AI模型训练对患者临床数据的依赖。
过去AI在临床反应预测上效果一直不算好的最大原因,便是想要收集海量、连续临床反应数据实在是太难了。
从机制上来看,研究人员将药物生物标志物分为了源域(source domain)和目标域(target domain)。
源域表示和测试样本不同的领域,但是有丰富的监督信息,在这里可以理解为体外细胞验证的数据。
目标域是测试样本所在的领域,无标签或只有少量标签,也就是患者数据。
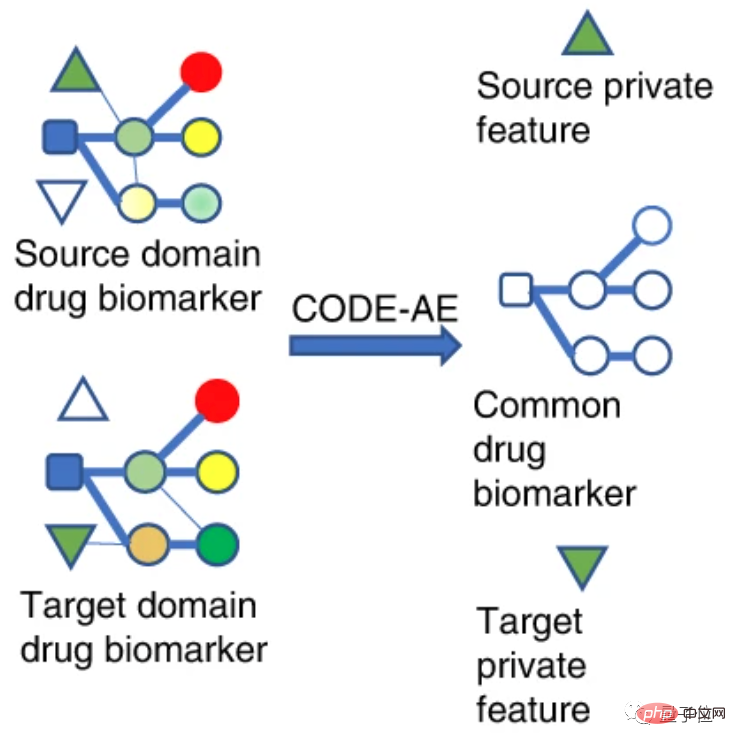
将不同领域的数据特征映射到同一个特征空间,使其在该空间中的距离尽可能近。
于是在特征空间中对源域训练的目标函数,就可以迁移到目标域,提高目标域上的准确率。
放在该研究背景下,源域和目标域都是药物生物标志物的数据特征,即药物靶标的数据特征。
具体来看模型框架,主要分为三个部分:预训练、微调和推理。
预训练主要用了自监督学习,构建一个特征编码模块,将体外细胞数据和患者数据的未标记基因表达谱,映射到嵌入空间中。这样一来可以把一些混杂因素排除掉,让两种数据的潜入分布一致,以消除系统偏差。
微调阶段,是在预训练的基础上再加一个监督模型,并利用已经标记的体外细胞数据来进行训练。
最后在推理阶段,先从预训练中获得的患者去歧对其嵌入,然后再利用调优后的模型,来预测患者对药物的反应。

在这种模式下,CODE-AE具备两个特点。
第一,它可以提取不连贯样本中的常见生物信号和私有表示,从而排除掉由于数据模式不同带来的干扰。
第二,将药物响应信号和混杂因素分离后,还可以实现局部对齐。
总结来看,CODE-AE可以理解为在标记和无标记数据的非相干数据模式嵌入空间中,选择唯一特征的过程。
为了论证模型的有效性,研究人员对9808位癌症患者的药物适用情况进行预测。
如果模型对患者情况预测出的位点结果,和他使用的药物靶点有关,就证明预测是正确的。
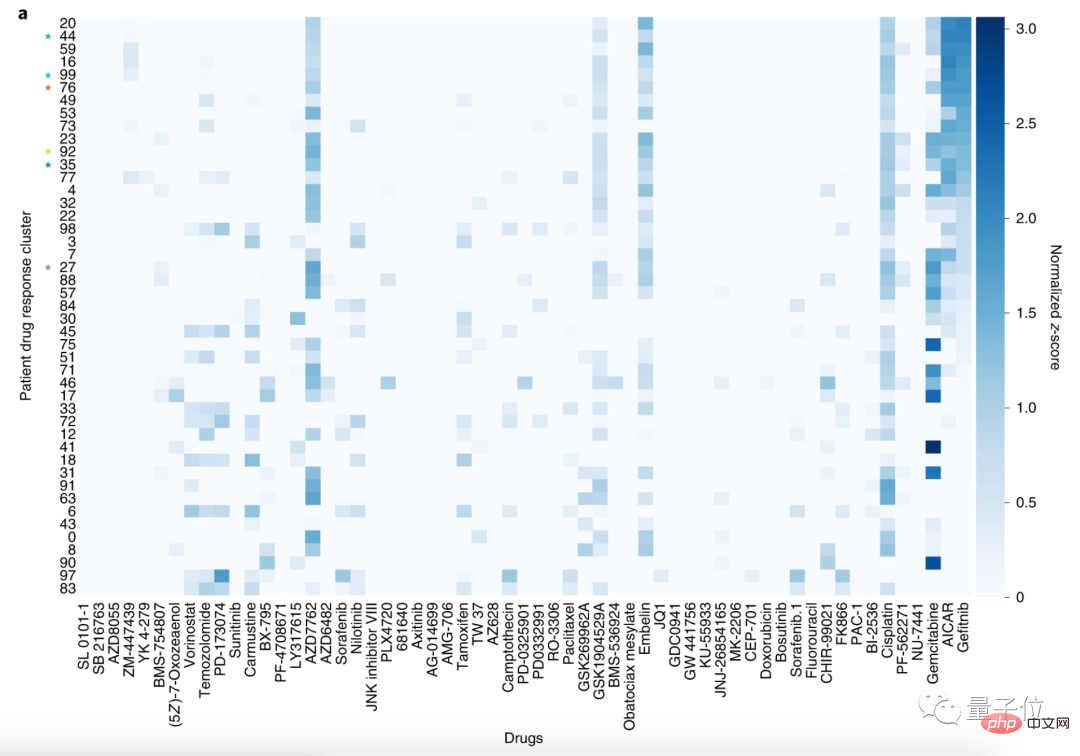
然后,研究人员将患者分为100个聚类,将59种药物也分为30个聚类。
通过这种分析方法,可以让具有相似药物反应谱的患者被分在一起。
在此,我们以肺鳞状细胞癌患者(LSCC)和非小细胞肺癌患者(NSCLC)的聚类为例。
在59种药物中,LSCC最敏感的药物为吉非替尼、AICAR和吉西他滨。
其中吉非替尼、AICAR的作用靶点都是一种表皮生长因子受体(EGFR),吉西他滨常被用于没有EGFR突变的非小细胞肺癌治疗。
论文表示,和这些药物作用模式一致,CODE-AE发现使用吉非替尼、AICAR的患者,药物反应图谱相似。
也就是说,CODE-AE发现了患者治疗的正确靶点,即可以预测适用药物。
如上研究团队来自纽约市立大学。
通讯作者为Lei Xie,他本科毕业于中国科学技术大学高分子物理专业。
硕士毕业于罗格斯大学计算机科学专业;博士同在罗格斯大学,但拿的是化学系学位。

据了解,该研究团队下一步将开发CODE-AE对新药临床反应在浓度、代谢方面的预测功能。
研究人员表示,该AI模型还有可能被调整为用于预测药物对人体的副作用影响。
值得一提的是,Nature子刊《Nature Machine Intelligence》专门关注人工智能和生命科学跨学科应用研究,每年收录论文平均数量在60篇左右。
论文地址:https://www.nature.com/articles/s42256-022-00541-0
参考链接:https://phys.org/news/2022-10-ai-accurately-human-response-drug.html
以上是华人团队成功开发AI预测癌症患者适用药物,成果发表在Nature子刊上的详细内容。更多信息请关注PHP中文网其他相关文章!

热AI工具

Undresser.AI Undress
人工智能驱动的应用程序,用于创建逼真的裸体照片

AI Clothes Remover
用于从照片中去除衣服的在线人工智能工具。

Undress AI Tool
免费脱衣服图片

Clothoff.io
AI脱衣机

AI Hentai Generator
免费生成ai无尽的。

热门文章

热工具

记事本++7.3.1
好用且免费的代码编辑器

SublimeText3汉化版
中文版,非常好用

禅工作室 13.0.1
功能强大的PHP集成开发环境

Dreamweaver CS6
视觉化网页开发工具

SublimeText3 Mac版
神级代码编辑软件(SublimeText3)

热门话题
 Debian邮件服务器防火墙配置技巧
Apr 13, 2025 am 11:42 AM
Debian邮件服务器防火墙配置技巧
Apr 13, 2025 am 11:42 AM
配置Debian邮件服务器的防火墙是确保服务器安全性的重要步骤。以下是几种常用的防火墙配置方法,包括iptables和firewalld的使用。使用iptables配置防火墙安装iptables(如果尚未安装):sudoapt-getupdatesudoapt-getinstalliptables查看当前iptables规则:sudoiptables-L配置
 Debian Apache日志级别如何设置
Apr 13, 2025 am 08:33 AM
Debian Apache日志级别如何设置
Apr 13, 2025 am 08:33 AM
本文介绍如何在Debian系统中调整ApacheWeb服务器的日志记录级别。通过修改配置文件,您可以控制Apache记录的日志信息的详细程度。方法一:修改主配置文件定位配置文件:Apache2.x的配置文件通常位于/etc/apache2/目录下,文件名可能是apache2.conf或httpd.conf,具体取决于您的安装方式。编辑配置文件:使用文本编辑器(例如nano)以root权限打开配置文件:sudonano/etc/apache2/apache2.conf
 如何优化debian readdir的性能
Apr 13, 2025 am 08:48 AM
如何优化debian readdir的性能
Apr 13, 2025 am 08:48 AM
在Debian系统中,readdir系统调用用于读取目录内容。如果其性能表现不佳,可尝试以下优化策略:精简目录文件数量:尽可能将大型目录拆分成多个小型目录,降低每次readdir调用处理的项目数量。启用目录内容缓存:构建缓存机制,定期或在目录内容变更时更新缓存,减少对readdir的频繁调用。内存缓存(如Memcached或Redis)或本地缓存(如文件或数据库)均可考虑。采用高效数据结构:如果自行实现目录遍历,选择更高效的数据结构(例如哈希表而非线性搜索)存储和访问目录信
 debian readdir如何实现文件排序
Apr 13, 2025 am 09:06 AM
debian readdir如何实现文件排序
Apr 13, 2025 am 09:06 AM
在Debian系统中,readdir函数用于读取目录内容,但其返回的顺序并非预先定义的。要对目录中的文件进行排序,需要先读取所有文件,再利用qsort函数进行排序。以下代码演示了如何在Debian系统中使用readdir和qsort对目录文件进行排序:#include#include#include#include//自定义比较函数,用于qsortintcompare(constvoid*a,constvoid*b){returnstrcmp(*(
 debian readdir如何与其他工具集成
Apr 13, 2025 am 09:42 AM
debian readdir如何与其他工具集成
Apr 13, 2025 am 09:42 AM
Debian系统中的readdir函数是用于读取目录内容的系统调用,常用于C语言编程。本文将介绍如何将readdir与其他工具集成,以增强其功能。方法一:C语言程序与管道结合首先,编写一个C程序调用readdir函数并输出结果:#include#include#includeintmain(intargc,char*argv[]){DIR*dir;structdirent*entry;if(argc!=2){
 Debian邮件服务器SSL证书安装方法
Apr 13, 2025 am 11:39 AM
Debian邮件服务器SSL证书安装方法
Apr 13, 2025 am 11:39 AM
在Debian邮件服务器上安装SSL证书的步骤如下:1.安装OpenSSL工具包首先,确保你的系统上已经安装了OpenSSL工具包。如果没有安装,可以使用以下命令进行安装:sudoapt-getupdatesudoapt-getinstallopenssl2.生成私钥和证书请求接下来,使用OpenSSL生成一个2048位的RSA私钥和一个证书请求(CSR):openss
 Debian OpenSSL如何防止中间人攻击
Apr 13, 2025 am 10:30 AM
Debian OpenSSL如何防止中间人攻击
Apr 13, 2025 am 10:30 AM
在Debian系统中,OpenSSL是一个重要的库,用于加密、解密和证书管理。为了防止中间人攻击(MITM),可以采取以下措施:使用HTTPS:确保所有网络请求使用HTTPS协议,而不是HTTP。HTTPS使用TLS(传输层安全协议)加密通信数据,确保数据在传输过程中不会被窃取或篡改。验证服务器证书:在客户端手动验证服务器证书,确保其可信。可以通过URLSession的委托方法来手动验证服务器
 Debian Hadoop日志管理怎么做
Apr 13, 2025 am 10:45 AM
Debian Hadoop日志管理怎么做
Apr 13, 2025 am 10:45 AM
在Debian上管理Hadoop日志,可以遵循以下步骤和最佳实践:日志聚合启用日志聚合:在yarn-site.xml文件中设置yarn.log-aggregation-enable为true,以启用日志聚合功能。配置日志保留策略:设置yarn.log-aggregation.retain-seconds来定义日志的保留时间,例如保留172800秒(2天)。指定日志存储路径:通过yarn.n






